:: Projekte :: DNAAnalyzer ::
DNA-Analyzer
Idee
Der DNA-Analyzer ist ein kleines Tool zur Analyse von DNA-Sequenzen. D.h. mit dem Tool ist es möglich die Häufigkeit von Basis-Sequenzen zu analysieren. Die Idee zu dem Tool kam mir bei der Lektüre des Buches
"Methoden der Bioinformatik - Eine Einführung" von Marc-Thorsten Hütt und Manuel Dehnert. In dem Buch wird die DNA-Sequenz aus den Roman "Jurassic Park" untersucht und nachgewiesen, daß es sich um keine reale DNA-Sequenz handeln kann. Die Idee hinter dem Nachweis ist der CG-Gehalt in einer DNA-Sequenz, der niedriger ist, als der Gehalt anderer Basenpaare. Beispielsweise sieht die Verteilung der Basenpaare des ersten menschlichen Chromosoms folgendermaßen aus:
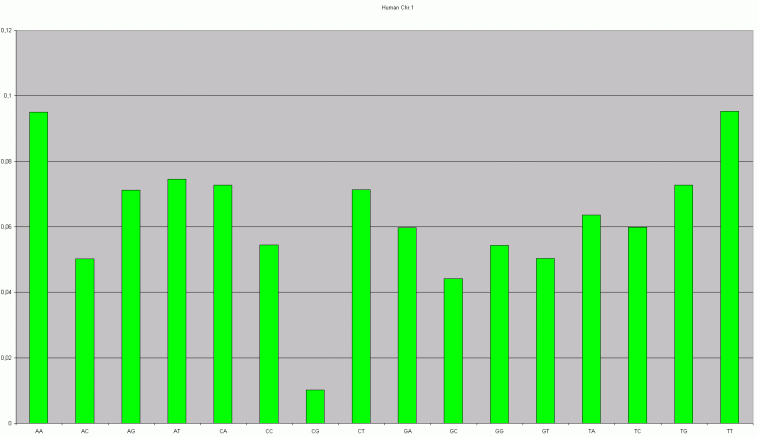
Dabei wurden die Basen A, C, G, T zu einer Sequenz der Länge zwei zusammengefaßt. Also: AA, AC, AG, AT, CA, CC, CG, CT, GA, GC, GG, GT, TA, TC, TG, TT. Man sieht, daß der CG-Gehalt deutlich niedriger ist, als der Gehalt der anderen Sequenzen.
Nimmt man jetzt zum Vergleich die Nullhypothese hinzu, dann erkennt man die Abweichungen, zum erwarteten Ergebnis noch deutlicher.
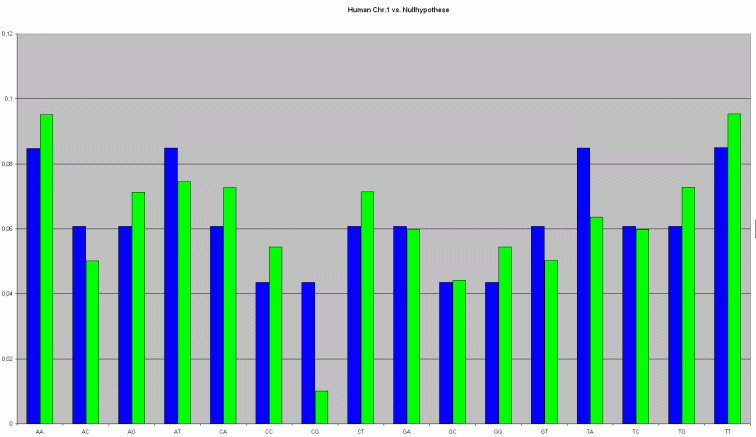
Man sieht jetzt ganz deutlich, daß die blauen Balken, die mit der Nullhypothese berechnete Wahrscheinlichkeit für die Basenpaare war, und die grünen Balken die Tatsächliche Verteilung ist.
Jurassic Park

Untersucht man jetzt die Sequenz aus den "Jurassic Park"-Buch erhält man ein völlig anderes Bild. Die untersuchte Sequenz sieht folgendermaßen aus:
>JurassicPark DinoDNA from the book Jurassic Park
gcgttgctgg cgtttttcca taggctccgc ccccctgacg agcatcacaa aaatcgacgc
ggtggcgaaa cccgacagga ctataaagat accaggcgtt tccccctgga agctccctcg
tgttccgacc ctgccgctta ccggatacct gtccgccttt ctcccttcgg gaagcgtggc
tgctcacgct gtaggtatct cagttcggtg taggtcgttc gctccaagct gggctgtgtg
ccgttcagcc cgaccgctgc gccttatccg gtaactatcg tcttgagtcc aacccggtaa
agtaggacag gtgccggcag cgctctgggt cattttcggc gaggaccgct ttcgctggag
atcggcctgt cgcttgcggt attcggaatc ttgcacgccc tcgctcaagc cttcgtcact
ccaaacgttt cggcgagaag caggccatta tcgccggcat ggcggccgac gcgctgggct
ggcgttcgcg acgcgaggct ggatggcctt ccccattatg attcttctcg cttccggcgg
cccgcgttgc aggccatgct gtccaggcag gtagatgacg accatcaggg acagcttcaa
cggctcttac cagcctaact tcgatcactg gaccgctgat cgtcacggcg atttatgccg
caagtcagag gtggcgaaac ccgacaagga ctataaagat accaggcgtt tcccctggaa
gcgctctcct gttccgaccc tgccgcttac cggatacctg tccgcctttc tcccttcggg
ctttctcatt gctcacgctg taggtatctc agttcggtgt aggtcgttcg ctccaagctg
acgaaccccc cgttcagccc gaccgctgcg ccttatccgg taactatcgt cttgagtcca
acacgactta acgggttggc atggattgta ggcgccgccc tataccttgt ctgcctcccc
gcggtgcatg gagccgggcc acctcgacct gaatggaagc cggcggcacc tcgctaacgg
ccaagaattg gagccaatca attcttgcgg agaactgtga atgcgcaaac caacccttgg
ccatcgcgtc cgccatctcc agcagccgca cgcggcgcat ctcgggcagc gttgggtcct
gcgcatgatc gtgctagcct gtcgttgagg acccggctag gctggcgggg ttgccttact
atgaatcacc gatacgcgag cgaacgtgaa gcgactgctg ctgcaaaacg tctgcgacct
atgaatggtc ttcggtttcc gtgtttcgta aagtctggaa acgcggaagt cagcgccctg
Faßt man die Basenpaare, wie beim menschlichen Chromosom, zusammen, dann erhält man folgende Verteilung:
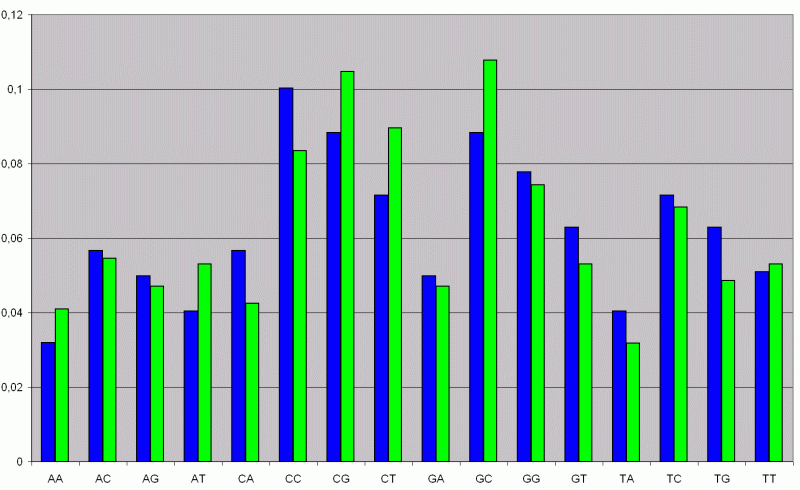
Man sieht sofort, daß der CG-Gehalt (grüner Balken) viel zu hoch ist. Er liegt sogar über dem der Nullhypothese (blauer Balken).
Das Lustige daran ist, daß Michael Crichton, auf Grund dieser Analyse für den zweiten Teil "The Lost World" den Wissenschaftler Dr. Mark Boguski eingeladen hat, der für ihn eine DNA-Sequenz erzeugte, die den oben gezeigten Makel nicht hat. Dr. Mark Boguski war auch derjenige, der den Fehler in der DNA gefunden hat und dazu ein Paper veröffentlicht hat. (Boguski, M.S. A Molecular Biologist Visits Jurassic Park. (1992) BioTechniques 12(5):668-669).
The Lost World

Das Ergebnis dieser Zusammenarbeit war folgende DNA-Sequenz:
>LostWorld DinoDNA from the book The Lost World
gaattccgga agcgagcaag agataagtcc tggcatcaga tacagttgga gataaggacg
gacgtgtggc agctcccgca gaggattcac tggaagtgca ttacctatcc catgggagcc
atggagttcg tggcgctggg ggggccggat gcgggctccc ccactccgtt ccctgatgaa
gccggagcct tcctggggct gggggggggc gagaggacgg aggcgggggg gctgctggcc
tcctaccccc cctcaggccg cgtgtccctg gtgccgtggg cagacacggg tactttgggg
accccccagt gggtgccgcc cgccacccaa atggagcccc cccactacct ggagctgctg
caaccccccc ggggcagccc cccccatccc tcctccgggc ccctactgcc actcagcagc
gggcccccac cctgcgaggc ccgtgagtgc gtcatggcca ggaagaactg cggagcgacg
gcaacgccgc tgtggcgccg ggacggcacc gggcattacc tgtgcaactg ggcctcagcc
tgcgggctct accaccgcct caacggccag aaccgcccgc tcatccgccc caaaaagcgc
ctgcgggtga gtaagcgcgc aggcacagtg tgcagccacg agcgtgaaaa ctgccagaca
tccaccacca ctctgtggcg tcgcagcccc atgggggacc ccgtctgcaa caacattcac
gcctgcggcc tctactacaa actgcaccaa gtgaaccgcc ccctcacgat gcgcaaagac
ggaatccaaa cccgaaaccg caaagtttcc tccaagggta aaaagcggcg ccccccgggg
gggggaaacc cctccgccac cgcgggaggg ggcgctccta tggggggagg gggggacccc
tctatgcccc ccccgccgcc ccccccggcc gccgcccccc ctcaaagcga cgctctgtac
gctctcggcc ccgtggtcct ttcgggccat tttctgccct ttggaaactc cggagggttt
tttggggggg gggcgggggg ttacacggcc cccccggggc tgagcccgca gatttaaata
ataactctga cgtgggcaag tgggccttgc tgagaagaca gtgtaacata ataatttgca
cctcggcaat tgcagagggt cgatctccac tttggacaca acagggctac tcggtaggac
cagataagca ctttgctccc tggactgaaa aagaaaggat ttatctgttt gcttcttgct
gacaaatccc tgtgaaaggt aaaagtcgga cacagcaatc gattatttct cgcctgtgtg
aaattactgt gaatattgta aatatatata tatatatata tatatctgta tagaacagcc
tcggaggcgg catggaccca gcgtagatca tgctggattt gtactgccgg aattc
Wenn man sich jetzt das Ergebnis der Analyse ansieht, fällt auf daß dieses mal der CG-Gehalt (grüner Balken) deutlich unter dem Wert der Nullhypothese (blauer Balken) liegt.
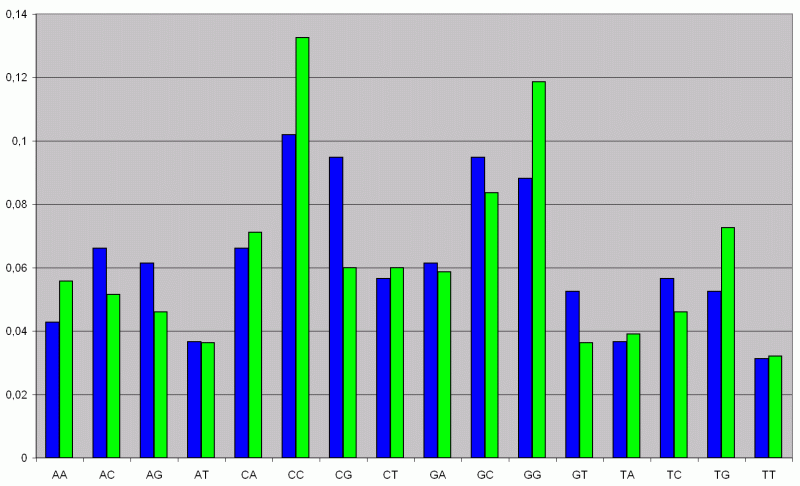
Programm
Das Programm analysiert die Häufigkeit von DNA-Sequenzen und vergleicht sie mit der Nullhypothese. D.h. es wird berechnet, welche Verteilung anhand des Vorkommens der einzelnen Basen A,C,G,T für die Sequenzen zu erwarten gewesen wären. Die Ergebnisse werden in eine Text-Datei geschrieben, die mit Tools wie Excel oder Mathematica eingelesen und als Balkendiagramm dargestellt werden können.
Das Programm wird folgendermaßen benutzt:
./DNAAnalyzer.exe DNADATEI SEQUENCELENGTH
Wobei
DNADATEI der Name der DNA-Datei ist. Normalerweise im Fasta-Format. Und
SEQUENCELENGTH die Länge der Basen-Sequenz. Also z.B. 2.
Für den Aufruf:
./DNAAnalyzer.exe Human_chr1_sequence.fa 2 erhält man als Ausgabe eine Text-Datei die folgendermaßen aussieht:
-----------------------------------
Number of Bases
A=64875254 ^= 29.1145
C=46493995 ^= 20.8654
G=46483769 ^= 20.8608
T=64974829 ^= 29.1592
-----------------------------------
AA 0.0847655 0.0950835
AC 0.0607487 0.0502142
AG 0.0607353 0.0712649
AT 0.0848956 0.0745729
CA 0.0607487 0.072721
CC 0.0435366 0.0544472
CG 0.0435271 0.0101329
CT 0.0608419 0.0713708
GA 0.0607353 0.059727
GC 0.0435271 0.0441625
GG 0.0435175 0.0543915
GT 0.0608286 0.0503362
TA 0.0848956 0.0636233
TC 0.0608419 0.0598128
TG 0.0608286 0.0728104
TT 0.0850259 0.0953289
Der obere Teil der Ausgabe enthält die Anzahl und die Wahrscheinlichkeit der einzelnen Basen. Im unteren Teil findet man in der ersten Spalte die Sequenz. In der zweiten Spalte die mit der Nullhypothese berechnete Wahrscheinlichkeit und in der dritten Spalte die tatsächliche Verteilung für die Sequenz.
Die Ausgabe ist so formatiert, daß sie mittels Copy&Paste direkt in Excel übernommen werden kann, um dort ein Balkendiagramm daraus zu erstellen.
Einschränkungen
Es ist maximal möglich Sequenzen der Länge 4 zu untersuchen und das Programm ist auf das Alphabet {A,C,G,T} beschränkt.
Download
Das Programm wurde mit dem GCC unter Cygwin übersetzt und benötigt deshalb die Cygwin-Dll, für die Ausführung. Diese Dll ist Bestandteil des ZIP-Pakets.
Download:
DNA-Analyzer





